In ParaView, the prefix "Para" stands for "parallel" : How can we activate parallelism in ParaView ?
How to use this tutorial
- This tutorial works only with the release 5.4.1 of ParaView
- If you have data to be visualized by ParaView, you can use your data.
- If you don’t have data yet, you can use some little scripts [23] written in python :
- first, copy the scripts to your personal disk space :
- When ParaView is started (see lower), you can read one script with the menu item :
Tools/Python shell/Run script
- first, copy the scripts to your personal disk space :
Starting a graphics session
ParaView must be used from a graphics session. Because we want to visualize very big datasets, we probably need more memory than the default. We must also specify that we want more than 1 core.
The complete story is here. You can use the following command to start a graphics session :
- using 25% of the graphics node’s cores
- using 25% of the graphics node’s memory
- with a comfortable display (if the width and the height of your workstation is sufficient)
Starting a visualization server
The ParaView gui [24] cannot use several cores. Only the readers and the visualization engines can use several cores. So we must start a server process, using mpi to make it to work in parallel. As we have 5 cores in this session, let’s dedicate 4 of them to server, keeping 1 core for the gui.
Open a terminal and type the following :
Starting the ParaView gui
Start ParaView version 5.4.1, using the main graphics menu : put the mouse over the root window, click with the right button, and choose the item Visualization_Applications/Paraview_5.4.1
Configuring the connection
The procedure described here should be done only the first time : the configured connection(s) will be kept from session to session.
- When Paraview is started, click on the menu item :
File/connect - A window entitled
Choose Server Configurationopens, click the buttonAdd Server - A window entitled
Edit Server Configurationopens with several fields, all fields being already filled. Modify the value of the field Name, writing the name you choose for the connection : saymy_pvserver, then click the buttonConfigure - A window entitled
Edit Server Launch Configurationopens, just click the buttonSave
Connecting to the pvserver
If not already done, start Paraview and click the menu item : File/connect, then select the my_pvserver line and click the Connect button. You are now connected to the pvserver, as  indicated in the Pipeline browser window (see figure). Besides, if you look at the window from which you launched pwserver, you should see this message :
indicated in the Pipeline browser window (see figure). Besides, if you look at the window from which you launched pwserver, you should see this message :
Loading and displaying data
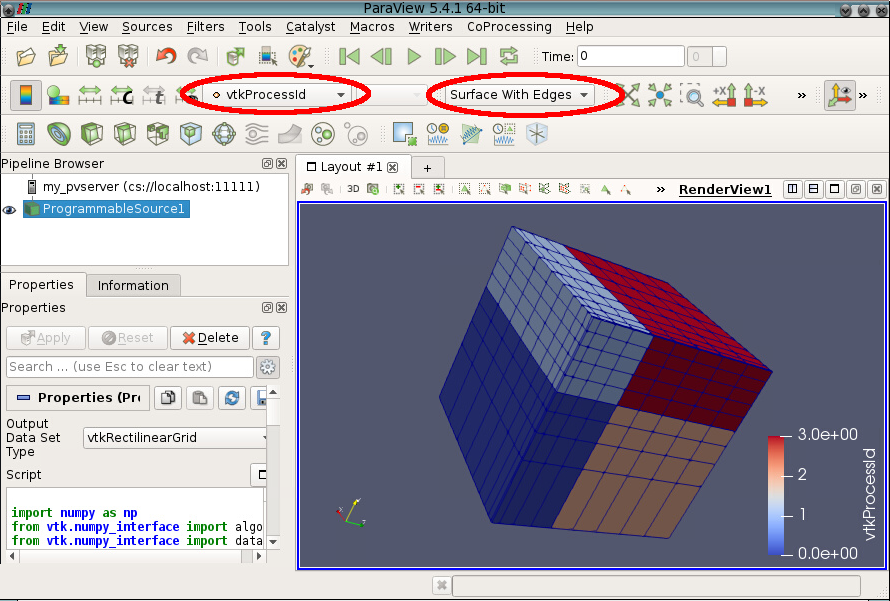
- Figure obtained with 4 processors, using the python script rectilinear.py
You can now load your datafiles, your state files, your python scripts, exactly as usual.
Warning : to read a python script, you must use the menu item : Tools/Python shell/Run script.
Data will be read by pvserver, and rended and displayed by ParaView. You should have a new property available with your data : vtkProcessId
You can easily display the domain decomposition made pvserver for reading your data (see the figure).
If you execute a filter, it will be executed by pvserver, thus using several cores, which can lead to faster execution if you have a huge quantity of data to treat
